I skal nu lave et Matlab GUI, som i stil med en tidligere opgave, kan segmentere en hjernetumor ved at bruge et "Remote ROI". I modsætning til tidligere, skal I nu segmentere samtlige skannede snit i et GUI. Jeg har lavet en del af forarbejdet for jer, men efterladt en række huller hist og her, som I selv skal udfylde.
Hent denne zip-fil, som indeholder dicom billeder og næsten færdige, præ-definerede funktioner. Gem den et passende sted, og åben .fig filen med guide.
pushbuttonLoadDicom
Denne funktion skal hente alle dicom billeder i et bibliotek. Jeg har overladt et par opgaver i callbacket til jer:
- Find callback'et til pushbuttonLoadDicom
- Indsæt en kommando til at indlæse et bibliotek ved den første linie markeret med % *** - outputtet skal gemmes i variablen dirnavn
- Skriv kommandoen til at gemme GUI data ved anden % *** markering
- displayImages er ikke programmeret endnu - derfor er den markeret med tredie % *** markering - denne markering skal fjernes når funktionen er klar.
Nu skal selve loadDicom funktionen gøres færdig:
- Øverste del af funktionen gentager i det store og hele funktionaliteten fra jeres tidligere opgave - den viser dicom filer i dirnavnet og indlæser hver enkelt. Processen monitoreres med en waitbar. Endelig gemmes billederne, som skaleres til at ligge mellem 0 og 1 i handles.Tumor variablen og dicominfo indlæses for at kunne beregne opløsningen samt voxel-størrelsen.
- Nederste del af funktionen mangler, og skal programmeres af jer:
- Først skal tre egenskaber på sliderImPos sættes
- Max værdien skal sættes til antallet af filer (numfiler)
- Brug set funktionen hertil:
- set(handles.sliderImPos,'Max',værdi)
- Brug set funktionen hertil:
- Den aktuelle værdi skal nulstilles til 1, dvs. egenskaben Value skal sættes til 1
- Sliderstep egenskaben skal sættes - den styrer hvor meget du flytter slideren når du klikker op/ned eller klikker i selve slideren. Den kaldes med to værdier: [KlikPåPilVærdi KlikISliderVærdi]. Værdierne er procentuelle, dvs. angiver hvor mange procent den skal flytte sig ved klik.
- KlikPåPilVærdien skal sættes til 1/(numfiler-1)
- KlikISliderVærdien skal sættes til 5/(numfiler-1)
- Max værdien skal sættes til antallet af filer (numfiler)
- Dernæst skal teksten til textImPos angives
- Brug set funktionen kombineret med sprintf til fx at skrive 1/20, 5/20, 10/20 etc.
- Først skal tre egenskaber på sliderImPos sættes
Gem dit arbejde, og tjek at funktionen virker efter hensigten, og kan vise en fil-dialog, hvor den efterspørger dicomfils-biblioteket samt viser en waitbar mens den henter billederne. Tjek også at textImPos ændres når du klikker op/ned på slideren.
displayImages
Denne funktion skal vise det aktuelle dicom billede. Initielt skal funktionen bestå af tre linier:
- Vælg axesDicom med funktionen axes
- Hent den afrundede værdi fra sliderImPos og gem den i variablen ImPos
- Vis det aktuelle billede (alle rækker, alle kolonner, ImPos) fra handles.Tumor.Im med funktionen imshow
Det var alt for nu - der kommer mere til senere, hvor man skal kunne vælge om man vil se det rå billede eller det segmenterede. Vend tilbage til callbacket for pushbuttonLoadDicom og fjern udkommenteringen af den tredie % *** markering.
Gem dit arbejde og prøv om det hele virker - du skulle nu kunne hente billeder og bladre rundt i dem med slideren.
SegmentBrain
Denne funktion segmenterer hjernen. I første omgang findes alle pixels over et threshold, dernæst bruges en morfologisk open opration for at fjerne tynde/små områder som hjerneskal og støj. Dernæst følger en connectivitetsanalyse, hvor kun det største sammenhængende område bevares. Dette ganges på de oprindelige billeder, hvorved en stak af hjernen i gråtoner bibeholdes. Det hele samles i en struktur, som er velegnet til en montage - et samlet plot af alle billederne i en stak på en gang.
Instruktioner:
- Indsæt først et funktionskald til funktionen SegmentBrain i callbacket til pushbuttonHjerneSegmentering
- SegmentBrain modtager handles som input argument, og skal også lægge værdier tilbage i handles.
- Efterfølg dette funktionskald med at gemme dine GUI data med den sædvanlige kommando.
- Find alle pixels, som overstiger værdien i editThreshold feltet. Gem deres indeks i variablen idx
- Dan et string element af typen disk med størrelsen i feltet editSELSize
- Udfør en morfologisk open opration med det dannede string element (sel) på bw variablen.
Dernæst følger en connectivitets undersøgelse. Sammenhængende områder inddeles i hver sin celle. Det største område bibeholdes, resten slettes. Derved forbliver kun områder, som hænger sammen med hjernen. Se yderligere forklaring i SegmentBrain.m
- Nu skal et der dannes en montage; et samlet plot af alle de segmenterede hjernesnit udført med en enkelt kommando.
- Først dannes selve datastrukturen, som skal ordnes på formen højde x bredde x 1 x snit
- Størrelsen 1 indikerer et gråtone billede. Havde det været farvebilleder skulle dette have været 3 (RGB).
- montageIm skal være produktet af hjernemasken ganget på de oprindelige billeder, som blev gemt i handles.Tumor i loadDicom funktionen - husk at bruge elementvise produkter.
- For at det vil virke, skal både masken og billederne have samme datatype - i dette tilfælde skal masken konverteres til double - fx double(maske)
- Vælg aksen axesMontage
- Plot hele stakken i variablen montageIm med funktionen montage.
Gem dit arbejde og prøv funktionen af. Prøv at justere tærskel og SEL str og iagttag ændringerne. NB: De prædefinerede værdier er ikke optimale! Find selv nogle, som virker bedre.
MarkNormal
Nu skal du programmere koden til knappen pushbuttonMarkNormal. Med den skal brugeren kunne udpege et normalt hjerneområde på det aktuelle dicombillede. Mean og standard deviation skal beregnes på det markerede område, som skal gemmes i handles.Tumor variablen:
- Gem det aktuelle snit i handles.Tumor.Im i variablen im:
- Alle rækker, alle kolonner, den afrundede værdi af sliderImPos
- Vælg axesDicom aksen
Nu følger selve roi funktionen. Her benyttes impoly, hvor brugeren skal klikke sig frem til et ROI, punkt for punkt. Dobbeltklik afslutter ROIet. wait(roi) sørger for, at funktionen først fortsætter, når brugeren har dobbeltklikket inde i sit ROI, for at markere, at normal området er færdigsegmenteret. Nu dannes en normal maske og roi'et slettes igen.
- Udregn nu middelværdi og standard deviation på idx pixels i im variablen og gem dem i handles.Tumor variablen.
- Gem GUI data med kommandoen guidata(XXX,XXX)
SegmentTumor
Nu skal selve tumoren segmenteres - proceduren er langt hen ad vejen den samme som for hjernesegmenteringen herover.
Instruktioner:
- Find de pixels som er det elementvise produkt af hjernemasken og selve billederne, begge gemt i handles.Tumor i hhv SegmentBrain og loadDicom funktionerne. Kun de pixels som overstiger Mean + værdien fra editThresholdTumor konverteret til double med str2double funktionen ganget med SD. Husk at konvertere masken til double.
- Pseudokode:
- idx = find(hjernemaske .* Dicombilleder > mean + editThresholdTumor * SD);
- Dan et tomt billede (består udelukkende af nuller) af samme størrelse som hjernemasken
- Dan et string element af typen disk med størrelsen fra editSELSizeTumor
- Udfør en open operation på tumor variablen med sel
Nu følger atter en connectivitetsundersøgelse magen til ovenfor.
- Dan ImTumor som produktet af tumor og de oprindelige billeder.
- Husk at bruge elementvis multiplikation, samt at konvertere tumor til double.
- Vælg axesMontage og plot tumorMontage stakken heri
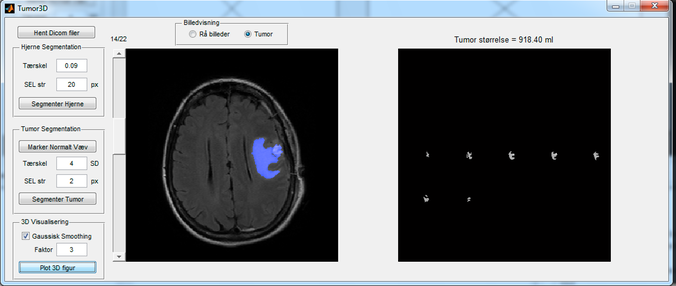
- Sæt titlen på dette plot til at være:
- Tumor størrelse = XXX.XX ml
- Brug title funktionen kombineret med sprintf
- Størrelsen kan udregnes som produktet af længden af tumor med voxelstørrelsen, som kan findes i handles.Tumor structet
Slutteligt dannes en farvestak af de oprindelige billeder, hvor den blå kanal modificeres, så tumoren bliver markeret med max-værdien, som er 1. Endelig vælges Tumor radiobutton over venstre akse (axesIm). Dette skal bruges for at få displayImages funktionen til at vise det farvede billede, hvor tumoren er blå, istedet for det oprindelige gråtone billede.
- Åbn funktionen displayImages.m
- Indsæt kode, som plotter det rå gråtonebillede, hvis radiobuttonIm er valgt i panelet uipanelImTumor (det over den venstre akse). Ellers skal det farvede billede med tumoren markeret med blå plottes
- Undersøg om det valgte tag i uipanelImTumor er radiobuttonIm:
- strcmp(get(get(handles.uipanelImTumor,'SelectedObject'),'Tag'),'radiobuttonIm')
- Hvis ja, plot det oprindelige billede med den funktion du allerede havde programmeret
- Ellers skal handles.Tumor.ImTumor vises med alle rækker, alle kolonner, alle farvekanaler og med ImPos snittet
- Brug imshow kommandoen
- Undersøg om det valgte tag i uipanelImTumor er radiobuttonIm:
Gem din funktion og prøv segmenteringen af - prøv at justere tærskel og SEL str, så kun selve tumoren markeres. NB: De prædefinerede værdier er ikke optimale! Find selv nogle, som virker bedre.
Plot3D
Nu mangler kun muligheden for at plotte de fundne data i 3D. Da I ikke har lært om 3D plots endnu, leveres funktionen as is. Dvs. den virker umiddelbart, og I skal ikke ændre i den. Vi gennemgår koden næste gang. Prøv den af og nyd synet af jeres arbejde i 3D.